Show the code
if(!require(blockrand)) install.packages("blockrand")https://www.equator-network.org/
独立随机分配,每组数量可能不等。
library(tidyverse)
# 假设需要 100 个样本,分为 4 个组
raw_data <- tibble(
include_order = 1:100
)
set.seed(20241011)
raw_data |>
mutate(简单assigned_group = sample(1:4, size = 100, replace = TRUE),
区组BRassigned_group = sample(rep(1:4, each=25))
) -> raw_data
table(raw_data$简单assigned_group)
#>
#> 1 2 3 4
#> 24 24 27 25
table(raw_data$区组BRassigned_group)
#>
#> 1 2 3 4
#> 25 25 25 25
raw_data
#> # A tibble: 100 × 3
#> include_order 简单assigned_group 区组BRassigned_group
#> <int> <int> <int>
#> 1 1 3 3
#> 2 2 2 1
#> 3 3 2 2
#> 4 4 4 4
#> 5 5 1 4
#> 6 6 1 1
#> 7 7 2 2
#> 8 8 3 1
#> 9 9 2 3
#> 10 10 1 1
#> # ℹ 90 more rows完全独立分配无法保证样本量恰好相等。如果是针对受试者的分组,可按照受试者的入组顺序,入组的第1名受试者进入组3,入组的第2名受试者分入组2,以此类推。
使用sample(rep(1:4, each=25))随机排列固定数量的标签保证各组数量相等,但分配过程不是独立的。实际上是一种区组随机化(当区组大小为100,即一个区组包含所有样本,且按1:1:1:1分配),它保证了每组数量相等,但破坏了受试者之间的独立性(因为每个组的数量固定,分配具有依赖性)。
在临床试验中,小样本情况下通常推荐使用限制性随机化以保证组间均衡
区组随机化:通过将受试者分成固定大小的区组,然后在每个区组内随机分配受试者到不同组别,确保在任何时候组间样本量接近相等。例如,使用大小为4的区组(每个区组内包含1,2,3,4各一次,随机排列),则每分配4个受试者,每个组恰好1例。这样,在100例中,仍然保证每组25例。
if(!require(blockrand)) install.packages("blockrand")library(blockrand)
# 使用区组随机化,指定区组大小为4 生成25个区组(每个区组4个,共100个)
set.seed(20241012)
# 创建区组随机化方案
blockrand_data <- blockrand(
n = 100, # 总样本量
num.levels = 4, # 分组数量
block.sizes = 1, # 区组大小
id.prefix = "ID", # 受试者ID前缀
block.prefix = "Block", # 区组ID前缀
stratum = "Center1" # 分层名称(单中心)
)
table(blockrand_data$treatment)
#>
#> A B C D
#> 25 25 25 25
blockrand_data
#> id stratum block.id block.size treatment
#> 1 ID001 Center1 Block01 4 B
#> 2 ID002 Center1 Block01 4 C
#> 3 ID003 Center1 Block01 4 A
#> 4 ID004 Center1 Block01 4 D
#> 5 ID005 Center1 Block02 4 D
#> 6 ID006 Center1 Block02 4 A
#> 7 ID007 Center1 Block02 4 C
#> 8 ID008 Center1 Block02 4 B
#> 9 ID009 Center1 Block03 4 D
#> 10 ID010 Center1 Block03 4 C
#> 11 ID011 Center1 Block03 4 B
#> 12 ID012 Center1 Block03 4 A
#> 13 ID013 Center1 Block04 4 B
#> 14 ID014 Center1 Block04 4 A
#> 15 ID015 Center1 Block04 4 C
#> 16 ID016 Center1 Block04 4 D
#> 17 ID017 Center1 Block05 4 D
#> 18 ID018 Center1 Block05 4 A
#> 19 ID019 Center1 Block05 4 C
#> 20 ID020 Center1 Block05 4 B
#> 21 ID021 Center1 Block06 4 C
#> 22 ID022 Center1 Block06 4 A
#> 23 ID023 Center1 Block06 4 B
#> 24 ID024 Center1 Block06 4 D
#> 25 ID025 Center1 Block07 4 A
#> 26 ID026 Center1 Block07 4 D
#> 27 ID027 Center1 Block07 4 C
#> 28 ID028 Center1 Block07 4 B
#> 29 ID029 Center1 Block08 4 C
#> 30 ID030 Center1 Block08 4 D
#> 31 ID031 Center1 Block08 4 A
#> 32 ID032 Center1 Block08 4 B
#> 33 ID033 Center1 Block09 4 C
#> 34 ID034 Center1 Block09 4 D
#> 35 ID035 Center1 Block09 4 B
#> 36 ID036 Center1 Block09 4 A
#> 37 ID037 Center1 Block10 4 C
#> 38 ID038 Center1 Block10 4 B
#> 39 ID039 Center1 Block10 4 D
#> 40 ID040 Center1 Block10 4 A
#> 41 ID041 Center1 Block11 4 B
#> 42 ID042 Center1 Block11 4 D
#> 43 ID043 Center1 Block11 4 A
#> 44 ID044 Center1 Block11 4 C
#> 45 ID045 Center1 Block12 4 C
#> 46 ID046 Center1 Block12 4 B
#> 47 ID047 Center1 Block12 4 A
#> 48 ID048 Center1 Block12 4 D
#> 49 ID049 Center1 Block13 4 A
#> 50 ID050 Center1 Block13 4 C
#> 51 ID051 Center1 Block13 4 B
#> 52 ID052 Center1 Block13 4 D
#> 53 ID053 Center1 Block14 4 B
#> 54 ID054 Center1 Block14 4 C
#> 55 ID055 Center1 Block14 4 A
#> 56 ID056 Center1 Block14 4 D
#> 57 ID057 Center1 Block15 4 A
#> 58 ID058 Center1 Block15 4 B
#> 59 ID059 Center1 Block15 4 C
#> 60 ID060 Center1 Block15 4 D
#> 61 ID061 Center1 Block16 4 D
#> 62 ID062 Center1 Block16 4 A
#> 63 ID063 Center1 Block16 4 B
#> 64 ID064 Center1 Block16 4 C
#> 65 ID065 Center1 Block17 4 C
#> 66 ID066 Center1 Block17 4 B
#> 67 ID067 Center1 Block17 4 D
#> 68 ID068 Center1 Block17 4 A
#> 69 ID069 Center1 Block18 4 A
#> 70 ID070 Center1 Block18 4 D
#> 71 ID071 Center1 Block18 4 C
#> 72 ID072 Center1 Block18 4 B
#> 73 ID073 Center1 Block19 4 C
#> 74 ID074 Center1 Block19 4 A
#> 75 ID075 Center1 Block19 4 D
#> 76 ID076 Center1 Block19 4 B
#> 77 ID077 Center1 Block20 4 A
#> 78 ID078 Center1 Block20 4 D
#> 79 ID079 Center1 Block20 4 B
#> 80 ID080 Center1 Block20 4 C
#> 81 ID081 Center1 Block21 4 B
#> 82 ID082 Center1 Block21 4 C
#> 83 ID083 Center1 Block21 4 A
#> 84 ID084 Center1 Block21 4 D
#> 85 ID085 Center1 Block22 4 B
#> 86 ID086 Center1 Block22 4 C
#> 87 ID087 Center1 Block22 4 A
#> 88 ID088 Center1 Block22 4 D
#> 89 ID089 Center1 Block23 4 B
#> 90 ID090 Center1 Block23 4 C
#> 91 ID091 Center1 Block23 4 A
#> 92 ID092 Center1 Block23 4 D
#> 93 ID093 Center1 Block24 4 C
#> 94 ID094 Center1 Block24 4 A
#> 95 ID095 Center1 Block24 4 B
#> 96 ID096 Center1 Block24 4 D
#> 97 ID097 Center1 Block25 4 A
#> 98 ID098 Center1 Block25 4 C
#> 99 ID099 Center1 Block25 4 B
#> 100 ID100 Center1 Block25 4 D
# 检查区组分布
blockrand_data |> group_by(block.id) |>
summarise(
groups = paste(sort(unique(treatment)), collapse = ","),
n = n()
)
#> # A tibble: 25 × 3
#> block.id groups n
#> <fct> <chr> <int>
#> 1 Block01 A,B,C,D 4
#> 2 Block02 A,B,C,D 4
#> 3 Block03 A,B,C,D 4
#> 4 Block04 A,B,C,D 4
#> 5 Block05 A,B,C,D 4
#> 6 Block06 A,B,C,D 4
#> 7 Block07 A,B,C,D 4
#> 8 Block08 A,B,C,D 4
#> 9 Block09 A,B,C,D 4
#> 10 Block10 A,B,C,D 4
#> # ℹ 15 more rows区组随机化的问题
根据不同的因素(性别)对受试者进行分组。如果性别是选定因素,则层数是二,对每层应用随机分组。这种形式的随机化可以减少治疗组中的特征不平衡,并提高统计功效。这种方法可以更容易地为不同的预后因素生成区组随机化列表。
分层随机化的问题
分层区组随机化分组
多中心临床试验中普遍采用的方法是以中心分层,然后在各中心内进行区组随机化,即称为分层的区组随机化。
动态随机化是指在临床试验的过程中每例患者分到各组的概率不是固定不变的,而是根据一定的条件进行调整的方法,它能有效地保证各试验组间例数和某些重要的非处理因素接近一致。动态随机化包括瓮(urn)法、偏币(biased coin)法、最小化(minimization)法等。
最小化
最小化随机化的核心是通过动态平衡组内特征分配,使得组间的差异最小。
最小化被用作平衡临床试验预后因素的工具。第一例受试者通过简单随机化分配到一个治疗组,然后根据先前的受试者及其在临床试验中的安置分配其余受试者,以平衡预后因素。目的是克服分层随机化的挑战。
# 加载randomizeR包
library(randomizeR)最小化的问题
在分配前对受试者的治疗组分配保密。这有助于减少选择偏倚,防止研究人员影响受试者分组。
首先要求产生随机分配序列和确定受试对象合格性的研究人员不应该是同一个人
其次,如果可能,产生和保存随机分配序列的人员最好是不参与试验的人员
按顺序编码、不透光、密封的信封(sequentially numbered,opaque, sealed envelopes )这是最常用的一种方法。每个研究对象所接受的治疗方案由产生的随机分配序列产生,并被放入按顺序、密封、不透光的信封中。合格的受试对象同意进入试验时,信封才能被打开,受试对象才能接受相应的处理措施。但是这种方法有一定的局限性。(1)如果研究者在试验前提前打开信封而将患者分配至预期的治疗组,将会破坏了研究的随机性。(2)如果将信封对着极强的光线,有时信封里面的内容能被看见。因此,为了避免这些问题,目前在临床研究中比较常用的做法是用来无碳复写纸按顺序、密封、不透光的信封。一旦无碳复写纸被开启后,就无法还原。这样,就比较容易发现研究者是否提前知道随机分配序列。信封法适用于单中心小样本的临床研究。
中心随机( central randomisation)系统为了避免按顺序、密封、不透光的信封所带来的问题,常用的一种方法是“远程”随机。“远程随机”是指当研究人员确定合格的研究对象后,通过电话或者网络将患者的基本信息传递给中心随机系统,然后获得每个患者的治疗分配方案。从今后的发展趋势来看,这种“远程”的中心随机系统将会取代信封系统。中心随机法适用于大型多中心研究。
随机分配方案隐匿与盲法
常与盲法相混淆。随机分配方案的隐匿是指通过在随机分配时防止随机序列被事先知道,而避免选择性偏倚;它在临床试验最后一名患者完成分组后即告结束;而盲法是为了避免干预措施实施过程中和结局指标测量时来自受试者和研究者的主观偏性,盲法需要在整个治疗和随访过程中保持盲的状态,直到试验干预和结局测量完成后才结束。盲法并非是在所有的临床试验中都能进行,但是随机分配方案的隐匿却在任何临床试验中都能进行,无论是分配前或者在分配的时点时。例如,比较针灸和药物两种疗法治疗某种疾病的疗效,盲法是难以实施的,而随机分配方案的隐匿却是可行的。
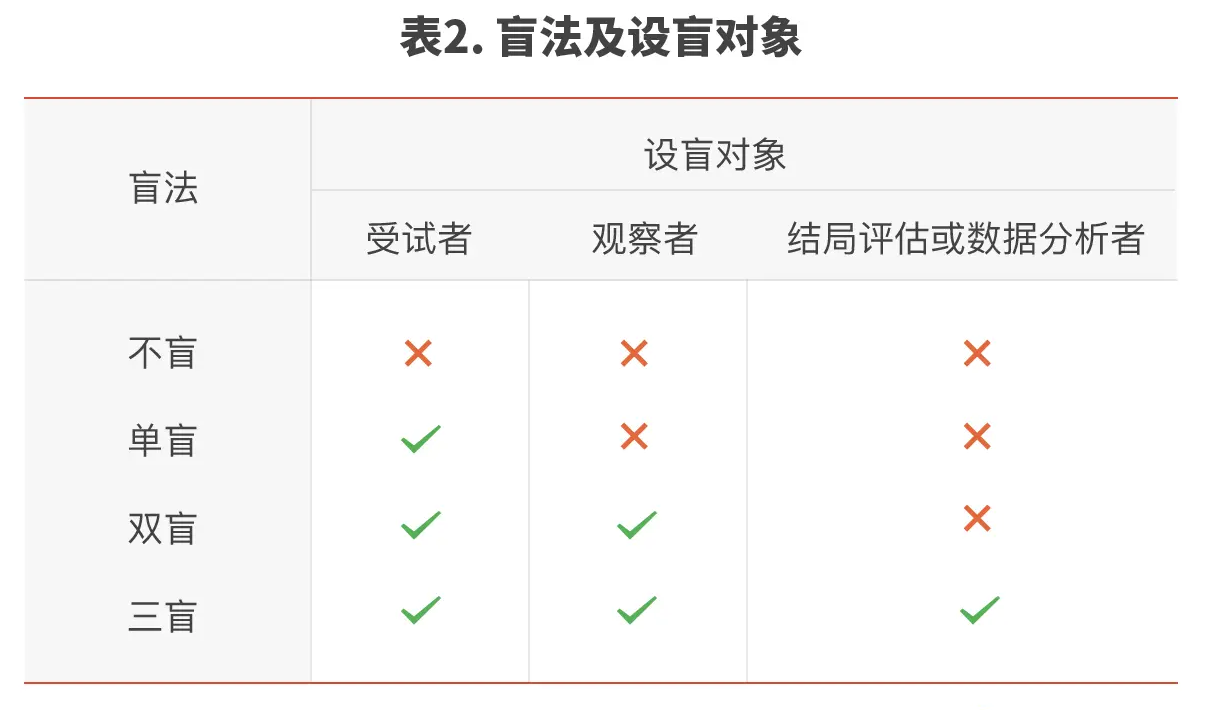
盲法是RCT的基本原则之一,但并不是所有的研究都必须采用或均能实行。比如比较某种手术与某药物治疗肺癌的效果,就不必要,也没有条件实施盲法。在这种情况下,只能进行开放试验,即研究对象和研究者均知道试验组和对照组的分组情况,试验公开进行。
在开放试验中,盲法无法实施,但是分组隐匿仍然可以实施。同时可以考虑对结局评价者、数据监察和统计分析人员设盲。其优点是易于设计和实施,研究者了解分组情况,便于对研究对象及时作出处理,其主要缺点是容易产生偏倚。
单盲试验(single-blinded):单盲试验是仅研究者知道每个病人用药的具体内容,而病人不知道,单盲试验虽可以避免来自病人主观因素的偏倚,但仍未能防止来自研究者方面的影响。
双盲(double blinded)试验:单盲试验是仅研究者知道每个病人用药的具体内容,而病人不知道,单盲试验虽可以避免来自病人主观因素的偏倚,但仍未能防止来自研究者方面的影响。
在双盲实施过程中,可能会出现一种情况:假设本研究中006号研究对象在研究过程中出现了严重的不良反应,要分析原因以便采取对策,此时必须揭盲。揭盲后研究实施者就知道了006号研究对象在使用乙药。但是,研究实施者在分组时已经知道006号研究对象为2组,揭盲后便知道了所有2组研究对象均在使用乙药,这会出现一人揭盲人人揭盲的情况。要避免这种情况,严格而规范的双盲试验不是给每个研究对象一个组别,按组别发药;而是给每个研究对象一个编码,按编码发药。盲底则由专人统一保管。
要避免一人揭盲人人揭盲的情况,双盲的具体做法是:
raw_data |>
mutate(
blind_id = 1001:1100,
drug_id = 1001:1100,
) ->raw_data
raw_data
#> # A tibble: 100 × 5
#> include_order 简单assigned_group 区组BRassigned_group blind_id drug_id
#> <int> <int> <int> <int> <int>
#> 1 1 3 3 1001 1001
#> 2 2 2 1 1002 1002
#> 3 3 2 2 1003 1003
#> 4 4 4 4 1004 1004
#> 5 5 1 4 1005 1005
#> 6 6 1 1 1006 1006
#> 7 7 2 2 1007 1007
#> 8 8 3 1 1008 1008
#> 9 9 2 3 1009 1009
#> 10 10 1 1 1010 1010
#> # ℹ 90 more rows
# 给研究实施者提供
raw_data |> select(contains("id"))
#> # A tibble: 100 × 2
#> blind_id drug_id
#> <int> <int>
#> 1 1001 1001
#> 2 1002 1002
#> 3 1003 1003
#> 4 1004 1004
#> 5 1005 1005
#> 6 1006 1006
#> 7 1007 1007
#> 8 1008 1008
#> 9 1009 1009
#> 10 1010 1010
#> # ℹ 90 more rows同时,分发的药品包装上也有与设盲编码一一对应的编码。
在随机化分组完成后,001号研究对象将分入3组,002号将分入2组。研究设计者在编码为1001的药品包装内分装C药,在编码为1002的药品包装内分装乙药。同时仅给研究实施者提供研究对象序号、设盲编码和标有设盲编码的药品 ( Table 21.2)。
而盲底( Table 21.1 )则只有研究设计者知道,并由专人保管。此时,研究实施者按照顺序纳入研究对象后,仅给研究对象标有编码的药品即可。研究实施者和研究对象均不知道分组和用药情况。
这样做的优点是:出现严重不良反应需要揭盲时,只需个别地揭盲,不容易泄密。但需要注意的是,必须建立与健全专项的药品保管、领用和发放制度;领发药品的各个环节都要认真核对编码,防止出错。实际上,如此执行,也做到了分组隐匿。
需要注意的是,试验结束前盲底泄露或应急信件拆阅超过20%,双盲试验即告失败。
在双盲实施的时候,还有可能出现一种偏倚:数据监察和统计分析人员知道研究分组情况,可能会对数据有选择性的取舍,造成不真实的情况。如在双盲实施的过程中,对数据监察和统计分析人员也设盲,此为三盲。
在研究实施过程中,将所有的临床试验数据输入数据库并经过核查,确证准确无误将数据锁定后,才进行第一次揭盲,即分出A、B两组,但哪一组是试验组、哪一组是对照组并不清楚。之后进行统计分析,待A组和B组分析数据出来后,再第二次揭盲,即明确A、B两组分别代表试验组还是对照组。
盲法与前述分配隐匿的区别在于:分配隐匿主要控制一种选择偏倚,即控制研究实施者有倾向地选择研究对象进入试验组或对照组;而盲法除此之外,还能控制信息偏倚,即控制研究实施者、资料收集和数据分析者有倾向性的取舍研究结果。
空白对照 安慰剂(placebo)对照 阳性对照
有一定的重复观察样本, 一项研究不允许有太多的受试者失访, 在选择研究样本时一定要特别注意。
Cochrane 偏倚风险评估工具包括 7 个方面:
Jadad 量表在临床试验的评价工作中简单易行,具有一定的科学性,得到了许多学者的认可, 发表了大量以 Jadad 量表作为评价工具的系统评价, 其中相当一部分文献集中在药物临床试验为主的研究, 在非开放式 RCT 评价中发挥了重要作用,得到了大多数学者的认可。 但是,随着人们对临床试验实施标准的不断修改完善,尤其是开放式非药物临床试验的RCT 研究在临床上广泛开展,Jadad 量表的评价标准有时不能完全客观、透明地反映出开放式 RCT 的研究质量,从而可能将质量较高的 RCT 误认为低质量 RCT。